http://www.focus.de/gesundheit/gesundleben/vorsorge/risiko/bakterienschleuder-am-fuss-warum-sie-ihre-schuhe-an-der-tuer-ausziehen-sollten_id_4996972.html
Bakterienschleuder am FußMehr Keime als auf der Klobrille: Warum Sie die Schuhe an der Tür ausziehen sollten
Freitag, 09.10.2015, 13:49 · von FOCUS-Online-AutorinNicole M
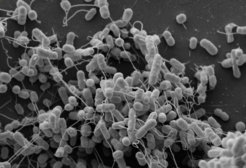
Bakterien lauern überall. Besonders gefürchtet sind sie auf öffentlichen Toiletten. Doch Studien zeigen: Unter den Schuhen kleben mehr Keime als auf einer Klobrille. Nach nur zwei Wochen tragen, tummeln sich mehr als 420.000 Bakterien auf unserem Schuhwerk.
- Schuhe sind hauptverantwortlich für die Verbreitung von Keimen.
- Auf 40 Prozent der Sohlen wimmelt es von Darmbakterien.
- Je ausgeprägter das Profil, desto mehr Keime bleiben hängen.
Die Japaner machen es richtig: Sie ziehen die Schuhe aus, bevor sie eine Wohnung betreten. In Deutschland streifen dagegen die wenigsten Menschen ihre Treter ab. Dabei wimmelt es auf Straßenschuhen nur so von unsichtbaren Krankheitserregern.
Eine mikrobiologischeStudie der University of Houstonbelegt, dass sich auf rund 40 Prozent der Sohlen auch das Bakterium Clostridium difficile (C. difficile) einnistet. Der Erreger ist einer der häufigsten Krankenhauskeime.
Eigentlich ist C. difficile ein harmloses Darmbakterium. Eine längere Einnahme vonAntibiotikakann jedoch seine Darmflora verändern oder sogar zerstören. Dann sorgen die Clostridien für erhebliche Beschwerden und verursachen unter Umständen sogareine Darmentzündung mit schweren Durchfällen.
Verbreitung durch Schuhe
Vier von zehn Menschen haben diese Erreger an ihren Sohlen kleben. Und das sind bei weitem nicht die einzigen Keime, die sie nach Hause schleppen. Zahllose Bakterien und Viren finden über das Schuhwerk ihren Weg in die Wohnung.
***********************
Selbsthilfe:
Versprühen Sie einfach TwinOxide ( Chlordioxidwasser) und Sie sind die Keime los, meine ich. ( W.St.)
***********************
Selbsthilfe:
Versprühen Sie einfach TwinOxide ( Chlordioxidwasser) und Sie sind die Keime los, meine ich. ( W.St.)
and the full-text about a product with chlorine dioxide in:
http://oru.diva-portal.org/smash/get/diva2:790688/FULLTEXT01.pdf